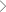近日,计算机科学技术学院生物信息研究组研发了微生物组局部比对算法Flex Meta-Storms(FMS),能够深入挖掘微生物组与其栖息环境之间隐含的联系。该成果于3月22在线日发表于生物信息学高水平期刊Bioinformatics (SCI IF=6.93)。该研究论文由青岛大学独立完成,计算机科学技术学院为第一单位,硕士研究生张明乾、张文科同学为共同第一作者,苏晓泉教授为通讯作者。该研究获得了国家重点研发计划、国家自然科学基金、泰山学者计划和山东省教育厅的支持,并获得软件著作权授权(登记号:2023SR0207831)。
微生物组与周围的环境有着密切的互作关系。Beta多样性是微生物组研究与应用的关键基础,能够定量评估复杂群落之间的差异。当前,beta多样性主要基于“全局比对”,即利用群落内全部成员计算总体距离(图1A)。然而,当环境特征(如某些疾病)只与一小部分的微生物有关时,这些微小的改变不足以对整体层面的距离产生影响,从而模糊了微生物与疾病之间的关联。
针对以上问题,苏晓泉教授前期提出了微生物组“局部比对”的概念,并应mSystems期刊(SCI IF= 7.324)主编邀请,将此算法概念发表于该期刊的“2021青年科学家特刊”。在此基础上,研究组首次对微生物组局部比对算法FMS进行了具体实现(图1B)。与现有的 “全局比对”算法相比,FMS能够识别出难以感知的细微差异,表现出了更好的敏感性和特异性。因此,FMS算法有助于深入理解微生物组与宿主的相互作用,促进微生物组大数据的深入研究与广泛应用。
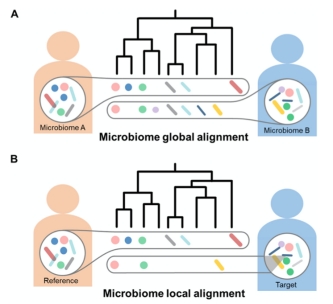
图1. 微生物组beta多样性距离计算的(A)全局比对模式与(B)局部比对模式
论文信息
1. Mingqian Zhang$, Wenke Zhang$, Yuzhu Chen, Jin Zhao, Shunyao Wu, Xiaoquan Su*, Flex Meta-Storms elucidates the microbiome local beta-diversity under specific phenotypes. Bioinformatics, 2023, DOI: https://doi.org/10.1093/bioinformatics/btad148.
2. Xiaoquan Su*, Elucidating the Beta-Diversity of the Microbiome: from Global Alignment to Local Alignment. mSystems, 2021, DOI: https://doi.org/10.1128/mSystems.00363-21.